第三回 利用CUBIC实现组织透明化与3D观察
![]()
东京大学研究生院医学系研究科系统药理学教室 洲㟢 悦生
◆前言
距今一百多年前,德国的Spalteholz开发出有机溶剂苯甲醇和水杨酸甲酯混合的组织透明化试剂,并报告了3D观察人体组织的案例1) (其中一部分样品至今仍在德累斯顿的公众卫生博物馆展览)。但在上世纪八十年代之前,组织透明化技术并未出现明显的发展与应用案例,直至在1989年,Dent提出关于Murray's clear或称为BABB法的改良Spalteholz试剂的应用实例报告2) ,并且在 九十年代以后俄罗斯的Tuchin等,台湾的Chiang等又提出了利用水溶性化合物进行的组织透明化方法3、4) ,开拓了近代组织透明化技术开发的道路。其中,以2007年的Dodt等5) ,2011年的滨等6)的荧光3D成像的使用案例为开端,以及随着近10年来数十种组织透明化方法的开发,组织透明化方法已经成为主要的组织观察手段之一。
我们团队于2014年发表了命名为CUBIC的全器官、全身级别细胞分析技术的概念7、8),并且不断开发高度组织透明化方法,以作为实现该概念的重要技术要素之一。本文将围绕其详情与应用实例进行介绍。
◆CUBIC技术概念与开发历程
CUBIC(Clear, Unobstructed Brain/Body Imaging Cocktails and Computational Analysis)是一项将整个器官,或全身所有细胞作为观察对象,无偏差且详尽地收集并分析细胞种类、细胞功能、细胞间的连接等信息的技术概念(图1)。
收集细胞分辨率的图像数据需要适合的光线模式,为了3D观察不透明的大型组织样品,必须使组织光学性透明。特别是在现实的时间尺度上,以整个器官与全身作为CUBIC的光学观察对象时,适合使用一种名为光片显微镜的特殊显微镜,通过在样品内产生光学切片并收集二维图像堆栈5) (图1、"整个器官的3D成像"部分)。
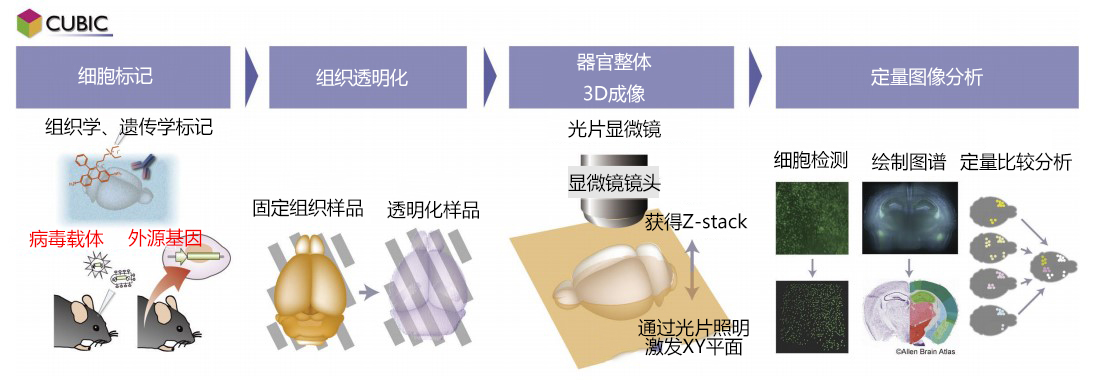
图1. CUBIC技术概念概略图
CUBIC是一项实现对整个器官和全身进行全面细胞分析的技术概念。将添加了荧光标记的组织样品光学性透明化,使用能够高速摄影大型样品整体的光片显微镜获取细胞分辨率的图像。根据所获图像的定量分析,提取细胞种类、细胞活动、细胞网络结构等信息。图谱图像来源Allen Brain Atlas(http://portal.brain-map.org)。
但是,由于光片显微镜从侧面照射激发光并从上方观察荧光信号,因此需将样品整体高度透明化(就如"透明"的字面意思几乎完全看不到的程度)。当然,荧光信号在透明化后仍需保留下来,以方便观察。
在笔者和研究人员开始开发CUBIC的2010年至2011年间,满足这些规范要求的组织透明技术尚未存在,因此需要开发新技术。恰逢理化学研究所的滨、宫胁等团队,发表了适合荧光蛋白信号保存的水溶性复合碱的组织透明化试剂"Scale"6),随即我们推出了独特的筛选方法,无需依赖直觉即可选择理想的水溶性化合物,并对40种化合物进行了定量筛选。 最后,成功开发了"ScaleCUBIC试剂",即在Scale的主要成分尿素中添加我们新发现的氨基醇 7) 。不仅如此,我们发现氨基醇能够去除组织中的主要吸光物质——血红素,并在后续 "脱色"过程中发挥重要作用8)。凭借这些特点,ScaleCUBIC试剂能够实现小鼠大脑、小鼠全身、人体组织等高度透明化并进行3D观察。
随后我们团队接连发表了观察小鼠全脑全细胞的膨润透明化法"CUBIC-X"、筛选1600种以上水溶性化合物后更新推出的"第二代 CUBIC试剂(CUBIC-L/R等)"、作为小鼠全脑分析基础的单细胞分辨率图谱 "CUBIC-Atlas"等的技术组成部分9-11)(表1)。
现在,我们更是准备发表3D染色方案、小鼠全脑单细胞分析基础、高速全细胞观察显微镜等新的基础技术。希望考虑使用CUBIC技术的各位用户也能继续关注这些研发成果。
|
试剂世代 |
功效 |
主要成分 |
文献 |
|
|
ScaleCUBIC-1 |
第1代 |
脱脂、脱色 |
氨基醇(四羟丙基乙二胺)、 尿素、Triton X-100 |
13) |
|
ScaleCUBIC-1A |
第1代 |
脱脂、脱色 |
氨基醇(四羟丙基乙二胺)、 尿素、Triton X-100 |
http://cubic.riken.jp/ |
|
ScaleCUBIC-2 |
第1代 |
调整折射率 |
氨基醇(三乙醇胺)、 尿素、蔗糖 |
13) |
|
CUBIC-X |
第1代 |
膨润+ 调整折射率 |
安替比林、咪唑 |
9) |
|
CUBIC-L |
第2代 |
脱脂、脱色 |
氨基醇(N-丁基二乙醇胺)、Triton X-100 |
11) |
|
CUBIC-P |
第2代 |
脱色 |
1-甲基咪唑、氨基醇(N-丁基二乙醇胺)、Triton X-100 |
11) |
|
CUBIC-B |
第2代 |
骨脱钙 |
EDTA、咪唑 |
11) |
|
CUBIC-HL |
第2代 |
脱脂、脱色 (部分人组织) |
1,3-双(氨基甲基)环己烷、 十二烷基苯磺酸钠 |
11) |
|
CUBIC-R |
第2代 |
调整折射率 |
安替比林、 烟酰胺或N-甲基烟酰胺 |
11) |
表1. CUBIC试剂一览
第二代试剂未使用Scale的主要成分尿素,因此将其统一记录为"CUBIC-〇〇"。
◆利用CUBIC进行组织透明化
所谓物体的光学透明,就是在入射光进入内部后几乎直线射出,不发生散射或吸收。组织之所以不透明,就是因为组织中各种成分的光学性质不均一,使入射光强烈散射。除此之外还含有像血液中的血红素那样强烈吸收可见光的色素。因此,组织透明化就是要通过1)去除组织中的光散射和光吸收物质,2)均一化组织成分与周围溶剂成分的光学性质(尤其是折射率),这两个步骤来达成。CUBIC试剂中表面活性剂(Triton X-100)与氨基醇对于上述1)中的过程十分重要。而 ScaleCUBIC试剂中的蔗糖,第二代CUBIC试剂中的安替比林与烟酰胺衍生物则对于上述2)中的过程尤为重要。
折射率调节剂拥有接近蛋白等生物体内物质折射率1.5左右的高折射率,"透明化"便是通过更换为折射率调节剂得以完成的。然而,ScaleCUBIC-1试剂也通过氨基醇与尿素的组合实现了相对较高的折射率(1.43),因此单种试剂便可实现较为明显的透明化,对于不熟悉透明化方法或想要在短时间内透明化少量样品并用共聚焦显微镜或双光子显微镜观察的用户,建议先尝试使用该试剂。
更高度的透明化则推荐使用脱脂和脱色剂+折射率调整剂这两个步骤进行透明化。此外,骨组织还需要进一步脱钙。我们在筛选1600多种大型化合物时也将脱钙化合物作为筛选对象,向EDTA中加入咪唑的脱钙试剂(CUBIC-B)。由于包含人在内的灵长类动物组织比啮齿动物组织的脱脂难度更高,因此,通过同样的大规模筛选开发了拥有更强脱脂能力的试剂(CUBIC-HL)11) 。
CUBIC试剂虽然拥有出色的荧光蛋白(GFP变体、mCherry、tdTomato、mKate2等)保存性,但是在表达量极少的情况下也无法获得足够的信号。不仅如此,该试剂还与部分荧光蛋白不兼容。因此,我们建议使用实际样品评估残留荧光信号。
将样品最小化并缩短处理时间的同时,在室温而不是37°C下进行脱脂,可改善荧光信号。另外,我们也公布了提高ScaleCUBIC-1试剂荧光信号保存性的ScaleCUBIC-1A(Reagent-1A)的试剂成分表(http://cubic.riken.jp/)。我们建议您将其与新的CUBIC-L试剂一起使用。图2总结了组织透明化过程。组织透明化是一种应用于固定的组织样品的技术,必须根据使用方案选择适合的固定方法。
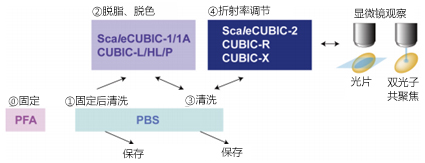
图2. 使用CUBIC的组织透明化和成像流程
CUBIC试剂针对多聚甲醛(PFA)固定组织进行了优化。固定组织经过脱脂、脱色后通过调整折射率实现透明化。中间的清洗过程可保存样品。试剂详情请参考表1。
在使用CUBIC时,通常推荐4%多聚甲醛进行固定。固定剂的pH值保持中性,且样品间的固定温度和交联时间原则上应保持一致。脱脂、脱色期间根据样品不同而有所区别,若是推荐实验方案中没有应用实例的组织请务必探讨实验条件。
由于脱脂、脱色后的样品呈现海绵状,需要进行防止样品损害的操作。对此,笔者团队是将样品放置在药勺上进行处理。此外,该阶段样品的长期保存可以使用含有防腐剂的PBS等。
由于折射率调节剂是浓度极高的试剂,需要提前替换成使用蒸馏水稀释为50%浓度的试剂处理1天以上后,再替换为100%浓度的试剂。假如稀释后的折射率调节剂的处理时间不足的话,可能导致内部无法透明化、组织变形等问题。观察后的组织通过使用PBS清洗,即可与脱脂、脱色后的样品在相同条件下进行保存。
◆CUBIC应用实例
医学生物学研究中组织透明化技术的应用实例逐年迅速增加,检索文献就可以查找到各种各样的生物种类以及组织的应用实例。例如在google scholar中搜索"CUBIC tissue clearing",即可发现8000多个匹配项和各种目标样品。其中还有鼠妇、蟹等甲壳类的适用案例12),另外,笔者意料之外的应用实例亦比比皆是。因此,考虑使用本试剂的用户,可先行搜索文献等寻找适用案例将会有助于研究的顺利开展。
接下来将介绍笔者团队关于CUBIC的应用实例。如上述所示,CUBIC将"整个器官和全身细胞的全面分析"作为概念,在开始的论文中7、13),首先提出了有无光刺激下两个个体在两种条件下的小鼠全脑神经活动比较分析的案例报告(图3A)。其后,还提出了将样品数量扩大,对给药后的小鼠进行时间顺序采样,分析其全脑神经活动的案例报
告14)。在该案例中,使用了总计8种条件下20只小鼠的大脑,最后进行在微阵列分析等方法中广泛应用的聚类分析,成功确定了特定时间段、实验条件下进行特异性活动的神经细胞群。感兴趣的读者可以参考原著论文。
另外,还有使用第二代CUBIC试剂使小鼠全身透明化,进行癌细胞转移模型小鼠全身分析的案例报告10)。从该案例的小鼠全身3D成像数据中可以看出,器官中四处分布的微小转移灶也可毫无遗漏地检测出来(图3),证明了CUBIC在全身分析研究中的实用性。
CUBIC还可以兼容核染色与免疫染色等组织学研究方法,并且部分抗体还可以将大脑、消化道、肾脏等器官整体染色并成像8、10、15)(图3C)。此外,还探讨了关于人组织与透明化、免疫染色、石蜡包埋术的相容性,并报告了CUBIC能够将3D病理学案例中的结肠癌淋巴结转移检测率提高至100% 16)。
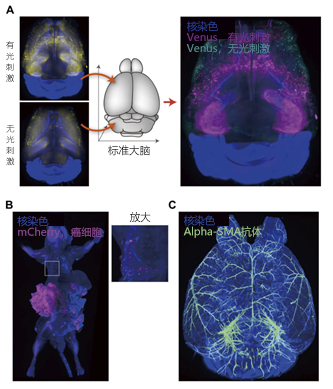
图3. CUBIC的应用案例
A:在光刺激后,对表达荧光蛋白Venus标记的转基因小鼠(Arc-dVenus Tg)19)的神经活动进行采样,然后与没有光刺激的对照组进行
A:比较。将两组数据嵌入标准大脑数据并进行标准化后,制作重合图像进行比较。使用数据改自文献13)。
B:癌细胞转移模型小鼠全身透明化,并进行3D成像的案例。能够毫无遗漏地检测出用mCherry标记的癌细胞的微小转移。使用数据改自文
A:献10)。
C:小鼠全脑免疫染色案例。使用标记血管平滑肌的α-Smooth Muscle Actin(SMA)抗体染色整个大脑后,进行透明化、3D成像。使用数
A:据改自文献10)。
在3D抗体染色时,建议使用抗体信噪比高且染色性好的单克隆抗体。笔者等人制备脱脂、脱色后的组织冷冻切片,确认抗体性能。
此外,为了不反复进行3D浸透过程,建议一抗使用直标荧光的抗体。根据抗体的不同,有些抗体会在折射率调节剂中剥离,建议在染色后用1%左右的PFA进行后续固定。但是,过强的固定会阻碍透明化,因此建议在保留抗体信号的最低限度下进行固定。
目前发表的实验方案仍未完全解决抗体的深层浸透问题,笔者等人正准备发表能够大大改善该问题的理想实验方案。感兴趣的用户请关注今后发表的论文。
◆结语
对于想要了解更多详情的用户,可以参考透明化、3D成像相关的英文综述17、18)。目前已经发表了数篇优秀的实验方案,用户可根据研究目的选择理想的实验方案。然而,包括我们研究团队在内的许多开发小组仍在不断地改进技术,希望各位不仅要关注早期的论文,更要关注新发布的研究文献。
若有CUBIC相关的技术问题需要咨询笔者(esusaki@m.u-tokyo.ac.jp),我们将随时为您解答。希望本项研究能够帮助到计划将组织透明化运用于自身研究中的各位用户。
◆参考文献
|
1) |
Spalteholz, W. : "Über das Durchsichtigmachen von menschlichen und tierischen Präparaten.", S. Hirzel, Leipzig (1914). |
|
2) |
Dent, J. A., Polson, A. G. and Klymkowsky, M. W. : "A whole-mount immunocytochemical analysis of the expression of the intermediate filament protein vimentin in Xenopus.", Development, 105, 61 (1989). |
|
3) |
Tuchin, V. V. et al. : Proc. SPIE, 3863, 10 (1999). |
|
4) |
Liu, Y.-C. and Chiang, A.-S. : "High-resolution confocal imaging and three-dimensional rendering.", Methods, 30, 86 (2003). |
|
5) |
Dodt, H. U. et al. : "Ultramicroscopy: Three-dimensional visualization of neuronal networks in the whole mouse brain.", Nat. Methods, 4, 331 (2007). |
|
6) |
Hama, H. et al. : "Scale: a chemical approach for fluorescence imaging and reconstruction of transparent mouse brain.", Nat. Neurosci., 14, 1481 (2011). |
|
7) |
Susaki, E. A. et al. : "Whole-brain imaging with single-cell resolution using chemical cocktails and computational analysis.", Cell, 157, 726 (2014). |
|
8) |
Tainaka, K. et al . : "Whole-body imaging with single-cell resolution by tissue decolorization.", Cell, 159, 911 (2014). |
|
9) |
Murakami, T. C. et al. : "A three-dimensional single-cell-resolution whole-brain atlas using CUBIC-X expansion microscopy and tissue clearing.", Nat. Neurosci., 21, 625 (2018). |
|
10) |
Kubota, S. I. et al. : "Whole-body profiling of cancer metastasis with single-cell resolution.", Cell Rep., 20, 236 (2017). |
|
11) |
Tainaka, K. et al. : "Chemical Landscape for Tissue Clearing based on Hydrophilic Reagents.", Cell Rep., 24, 2196 (2018). |
|
12) |
Konno, A. and Okazaki, S. : "Aqueous-based tissue clearing in crustaceans.", Zoological Lett., 4, 13 (2018). |
|
13) |
Susaki, E. A. et al . : "Advanced CUBIC protocols for whole-brain and whole-body clearing and imaging.", Nat. Protoc., 10, 1709 (2015). |
|
14) |
Tatsuki, F. et al. : "Involvement of Ca2+– Dependent Hyperpolarization in Sleep Duration in Mammals.", Neuron, 90, 70 (2016). |
|
15) |
Hasegawa, S. et al. : "Comprehensive three-dimensional analysis (CUBIC-kidney) visualizes abnormal renal sympathetic nerves after ischemia/reperfusion injury.", Kidney Int., 96, 129 (2019). |
|
16) |
Nojima, S. et al. : "CUBIC pathology: three-dimensional imaging for pathological diagnosis.", Sci. Rep., 7, 9269 (2017). |
|
17) |
Tainaka, K. et al. : "Chemical Principles in Tissue Clearing and Staining Protocols for Whole-Body Cell Profiling.", Annu. Rev. Cell Dev. Biol., 32, 713 (2016). |
|
18) |
Susaki, E. A. and Ueda, H. R. : "Whole-body and Whole-Organ Clearing and Imaging Techniques with Single-Cell Resolution: Toward Organism-Level Systems Biology in Mammals.", Cell Chem. Biol., 23, 137 (2016). |
|
19) |
Eguchi, M. and Yamaguchi, S. : "In vivo and in vitro visualization of gene expression dynamics over extensive areas of the brain.", Neuroimage, 44, 1274 (2009). |
